


-90252da93ac22e42.jpg)
Reportagem: Aline Tavares
Fotos: Marília Ruberti/Comunicação Butantan
Arte: Daniel das Neves
Nos anos 1960, uma bactéria extremamente resistente foi descoberta dentro das fontes termais do Parque Nacional de Yellowstone, nos Estados Unidos: a Thermus aquaticus. Naquele momento, não se sabia a importância que o microrganismo viria a ter na história da ciência – mais precisamente, uma enzima produzida por ele, a Taq polimerase. Mas foi essa proteína que, 20 anos depois, possibilitou aprimorar um dos métodos de diagnóstico mais rápidos e eficientes do mundo: a PCR (sigla em inglês de Reação em Cadeia da Polimerase). Não à toa, a revista Science a considerou a “molécula do ano” em 1989.
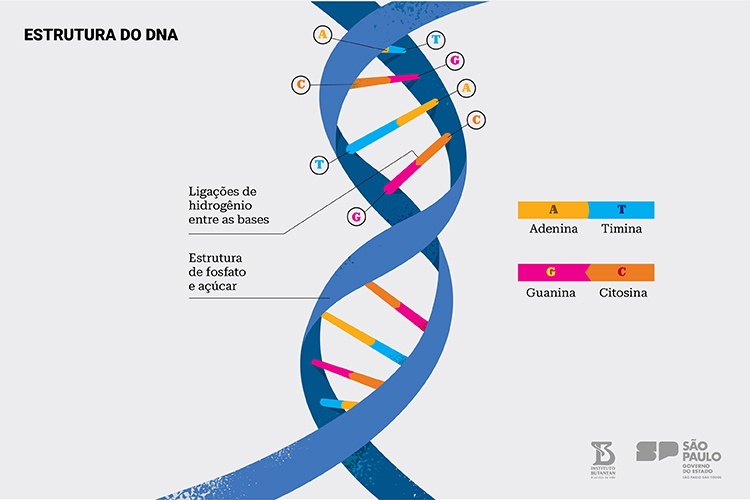
A técnica de PCR foi criada em 1983 pelo cientista norte-americano Kary Mullis. Na época, já se sabia que os seres vivos eram capazes de criar cópias de seu próprio DNA usando uma enzima chamada DNA polimerase. O DNA é uma molécula longa formada por duas fitas, conectadas por pequenos pares de moléculas chamadas nucleotídeos, que abrigam uma sequência genética. A duplicação do DNA é essencial para a vida, promovendo desde a cicatrização de um corte na pele até a formação de novas células e o crescimento dos organismos.
A PCR foi a tentativa de reproduzir essa atividade dentro do laboratório, usando a DNA polimerase para fazer uma reação em cadeia e criar múltiplas cópias de uma amostra de material genético, como se estivesse copiando as páginas de um livro. O problema é que a polimerase usada no passado era extraída da E. coli, uma bactéria frequentemente estudada nos laboratórios, mas que apresentava uma importante limitação, como conta o pesquisador científico do Laboratório de Bacteriologia do Instituto Butantan, Enéas de Carvalho.
“A enzima de E. coli não aguentava a alta temperatura necessária para conduzir a técnica e se degradava, exigindo que a cada ciclo fosse adicionada uma nova quantidade de enzima. A reação tinha que ser interrompida e reiniciada várias vezes, além de ser um processo totalmente manual – era preciso ficar colocando e tirando a amostra de banhos-maria”, explica.
A solução, curiosamente, estava presente na bactéria Thermus aquaticus: por viver em águas que atingiam temperaturas acima de 80°C, esse microrganismo é muito resistente ao calor – o que significa que a sua enzima também é. Apelidada de Taq polimerase, a proteína passou a ser usada na PCR em 1988 e revolucionou a técnica.

“Com o avanço da tecnologia e o desenvolvimento dos termocicladores, equipamentos que controlam a temperatura, todo o processo passou a ser automatizado e se tornou mais rápido”, diz Enéas. “A bactéria Thermus aquaticus já nem é mais necessária: o gene da Taq polimerase foi introduzido (clonado) na E. coli, de modo que ela agora produz a enzima da outra bactéria como se fosse dela. Hoje, a Taq polimerase é facilmente comercializada para laboratórios”, completa.
Uma das muitas funções da PCR é o diagnóstico de doenças infecciosas: a reação permite detectar a presença de genes de um vírus ou bactéria no organismo do paciente a partir da multiplicação do DNA de uma amostra de saliva, sangue ou secreção nasal, por exemplo, mesmo se a quantidade de material genético do patógeno for muito baixa – como quando a infecção está em fase inicial. Devido a essa alta sensibilidade, o teste é considerado padrão-ouro para identificar várias doenças.
Além do diagnóstico de enfermidades, a tecnologia pode servir para diversos fins, como testes de paternidade, investigação criminal, identificação de tumores, sequenciamento de DNA e clonagem.

O passo a passo da PCR
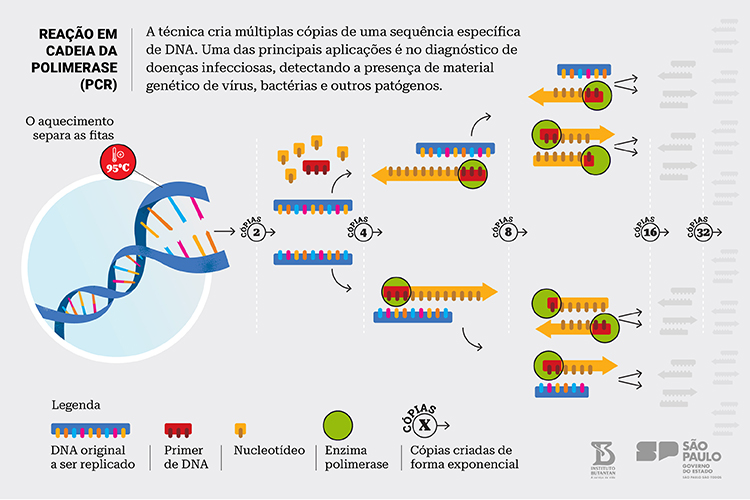
Para fazer a Reação em Cadeia da Polimerase, são necessários alguns ingredientes: a amostra de DNA que será analisada, a enzima DNA polimerase, os nucleotídeos e, por fim, os chamados primers (pedacinhos do DNA que se deseja detectar). Os primers servem para guiar o trabalho da enzima, como um “marcador de texto”, mostrando o trecho que ela deve copiar. Por exemplo: para um teste de influenza, os primers são produzidos com parte da sequência do vírus influenza, e só irão detectar o material genético daquele vírus.
“Consequentemente, apenas o material genético do vírus será copiado, não o DNA humano. O primer é encomendado sob medida pelos laboratórios para empresas especializadas em sintetizá-lo”, explica o pesquisador do Laboratório de Desenvolvimento e Inovação do Butantan, José Ricardo Jensen.
Com tudo isso em um tubinho, a amostra é aquecida a cerca de 95°C, processo necessário para separar as duas fitas do DNA e expor os nucleotídeos; então, a temperatura é reduzida gradativamente, permitindo que os primers grudem em cada uma das fitas no trecho de interesse; por fim, a polimerase fará as cópias, adicionando novos nucleotídeos e formando duas novas fitas, usando como molde o DNA alvo. Esses três passos são repetidos várias e várias vezes para criar quadrilhões de novas fitas.
Se a reação de fato acontecer e houver multiplicação do DNA, significa que existe material genético do vírus analisado na amostra – por isso um resultado positivo é descrito como “reagente”. Caso a reação da polimerase não aconteça, o resultado é negativo, e o laudo aponta a amostra do paciente como “não reagente”.
PCR em tempo real
A Reação em Cadeia da Polimerase foi sendo aprimorada ao longo dos anos, levando à criação da PCR em tempo real ou quantitativa (qPCR) em 2003. Enquanto a PCR convencional consiste em fazer as cópias do DNA durante um número fixo de ciclos e depois analisar o resultado (reagente ou não), a qPCR analisa e quantifica, em tempo real, a amplificação do DNA presente em uma amostra.
“Isso gera uma informação de dinâmica de amplificação, que é dependente da quantidade inicial de DNA na amostra. Ou seja, é possível determinar quanto de DNA viral havia na amostra inicial”, explica Enéas.
Para fazer a qPCR, são incluídas no processo moléculas fluorescentes que ajudam a identificar quanto de novo DNA está sendo formado. À medida que o material genético da amostra é copiado, a quantidade do DNA alvo aumenta exponencialmente, emitindo uma fluorescência que é detectada pelo equipamento. “Todo o processo ocorre dentro da máquina. O aparelho capta a fluorescência e mostra a quantidade do vírus em um gráfico”, diz José Ricardo.
Em situações de emergência, a identificação rápida é essencial para isolar os pacientes e conter a disseminação de um patógeno. Por isso, a tecnologia qPCR foi muito utilizada durante a pandemia de Covid-19, contribuindo para acelerar os diagnósticos e ajudar no controle da doença, liberando resultados em poucas horas. Uma das frentes de diagnóstico no estado de São Paulo foi coordenada pelo próprio Butantan, a Rede de Laboratórios para Diagnóstico do Coronavírus SARS-CoV-2, que realizava 20 mil testes diagnósticos por dia utilizando justamente a qPCR.

Respostas na natureza
Segundo o pesquisador Enéas de Carvalho, muitas atividades realizadas por cientistas são baseadas em tentar reproduzir o que a natureza faz. “Nós tiramos proveito de diversos fenômenos que observamos na natureza e trazemos para o laboratório como uma ferramenta de desenvolvimento tecnológico. E com a PCR foi assim”, conta.
Além da Taq polimerase, outra enzima amplamente utilizada na PCR é a Pfu, proteína extraída da arqueia Pyrococcus furiosus, um microrganismo proveniente da Ilha Vulcano, na Itália, descoberto em 1986 por pesquisadores alemães. Sua temperatura ideal é de nada menos que 100°C, o que o torna uma boa opção para técnicas que envolvem altas temperaturas.
Na PCR, enquanto a Taq é mais usada para diagnósticos, a Pfu tende a ser usada para clonagem, atividade que exige mais precisão. “A Taq é mais rápida e erra um nucleotídeo a cada 10 mil. Para diagnosticar doenças, é ideal pela agilidade e porque o objetivo é detectar a presença de um DNA de vírus ou bactéria – e eventuais erros de cópia não impactam nisso. Já a Pfu, embora demore mais, é recomendada para clonagem pois é mais precisa: sua taxa de erro é de um a cada 1,3 milhão de nucleotídeos”, esclarece Enéas.



